
GenomEast
GenomEast
RESPONSABLE DE PLATEFORME
Forts d'une expérience de plus de 20 ans, nous proposons une large gamme de services pour explorer les génomes, leur expression et leur régulation, du contrôle qualité des échantillons de départ jusqu'à l'analyse des données. Ces services sont destinés à l'ensemble de la communauté scientifique, nationale et internationale, publique et privée.



La plateforme est membre du noyau central de l'Infrastructure Nationale de Recherche France Génomique.
La plateforme est labellisée IBISA.
La plateforme est certifiée ISO 9001 - NF X 50-900.
Nos prestations
Nos prestations et nos recommandations sont détaillées dans les fiches produits ci-contre.
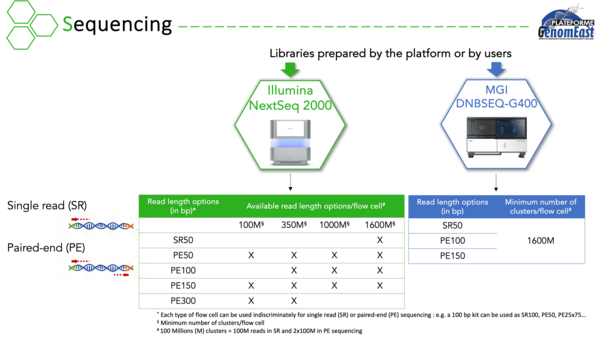
Actuellement équipés des séquenceur NextSeq 2000 Illumina et DNBSEQ-G400 de MGI, nous réalisons des projets d'analyse :
du transcriptome :
- RNA-seq
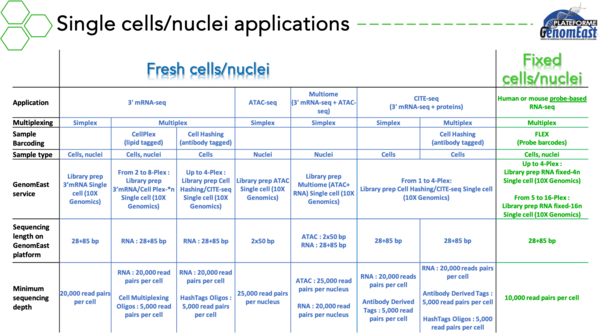
- Single cell RNA-seq (scRNA-seq)
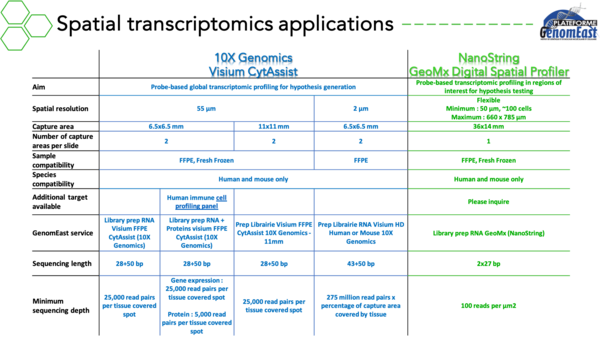
- Transcriptomique spatiale
de l'épigénome :
- ChIP-seq
- Cut & Run
- MeDIP-seq
- Nous avons également de l'expérience dans le séquençage de vos librairies ATAC-seq ou Cut & Tag
du génome :
- Régions ciblées du génome (exome, régions d'intérêts)
- Séquençage complet du génome
multiomique :
- Expression des gènes + ATAC en cellule unique
Pour ces projets, nous préparons et séquençons vos librairies en réalisant des contrôles qualité à chaque étape. Nous pouvons également réaliser l’analyse bioinformatique de vos données. Nous avons une expertise dans une grande variété d’applications et nous avons développé des pipelines bioinformatiques dédiés pour analyser les résultats correspondants.
Alternativement, vous pouvez utiliser notre capacité de séquençage pour séquencer vos propres libraires dans la mesure où elles sont compatibles avec la technologie Illumina.
Fiches produits
- Transcriptomique
- Epigénomique
Multiomique
- Génomique
Besoin d'aide pour le design de votre expérience ?
Consultez nos fiches produit ci-dessus ou nos schémas d’aide à la décision ci-après :
Librairies
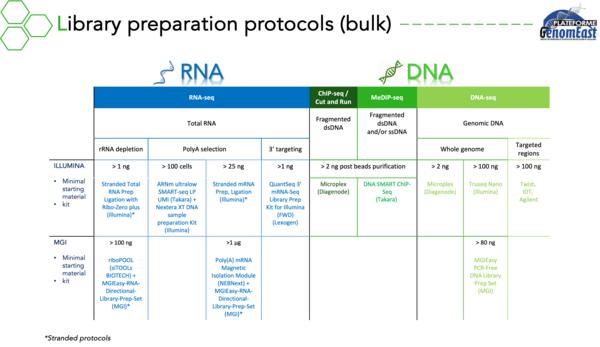
Cellules / Noyaux uniques
Transcriptomique spatiale
Séquençage
Des questions ?
N’hésitez pas à nous contacter (prenom.nom@igbmc.fr) :
Pour les projets portant sur l’ARN : Christelle Thibault et Céline Keime
Pour les projets portant sur l’ADN : Bernard Jost et Tao Ye
Soumettre un projet
Comment soumettre un projet à GenomEast ?
Via notre interface web
Quelle procédure suivre ?
Comment envoyer des échantillons à GenomEast ?
Tous nos services sont régis par les conditions générales de vente qui prévalent sur toutes les conditions d'achat, sauf accord écrit de notre part. Vous pouvez également consulter notre plan de gestion des données.
Formations
Nous partageons régulièrement nos connaissances et expertises à travers différents enseignements et formations, notamment :
L'UE « High Throughput Approaches » à l’Ecole Supérieure de Biotechnologie de Strasbourg (ESBS)
L’UE « Omique 2 » du Master 2 Biologie-Santé, parcours Recherche en Biomédecine à l’Université de Strasbourg
Le PhD Program de l’école Universitaire de Recherche IMCBio à Strasbourg
Le DU « Séquençage haut débit et maladies rares » à l'Université de Dijon
L'école de bioinformatique AVIESAN IFB Inserm
L’organisation de formations en bioinformatique pour le séquençage haut débit avec l’Inserm et le CNRS
Membres
Responsable de la plateforme
Christelle Thibault-Carpentier
Traitement des échantillons
- Bernard Jost (responsable)
- Marie Cerciat
- Amal El Amri
- Romain Kaiser
- Christelle Morgenthaler-Roth
- Angélique Pichot
- David Rodriguez
- Serge Vicaire
Bioinformatique
- Céline Keime (responsable)
- Matthieu Jung
- Stéphanie Le Gras
- Damien Plassard
- Tao Ye
Ressources
La plateforme est équipée de technologies de pointes parmis lesquelles :

Le séquenceur haut débit NextSeq 2000 (Illumina)

Le séquenceur haut débit AVITI (Element Biosciences)

La technologie en cellule unique du Chromium iX (10x Genomics)

La technologie spatial Visium CytAssist (10X Genomics)
Nos autres équipements:
- Séquençage haut débit : iSeq100 (Illumina)
- Transcriptomique spatiale : GeoMx Digital Spatial Profiler (NanoString)
- Contrôle qualité : Flash Reader Varioskan et Qubit Fluorometer (Thermo Fisher), 2100 Bioanalyzer et Fragment Analyzer AATI (Agilent)
- Sonicateur : E220 AFA system (Covaris)
- Infrastructure informatique : calcul (Dell) : 592 cœurs, stockage (Lenovo, GPFS) : 750 To
Réseaux et financements
France Génomique
Notre plateforme est partenaire du réseau France Génomique depuis la sélection initiale du projet dans le cadre des « Investissements d'avenir » en 2011. Ce réseau rassemble et partage les ressources des principales plateformes françaises de génomique et de bioinformatique.
Fondation maladies rares
Depuis 2011, nous sommes l’une des plateformes partenaires de la Fondation Maladies Rares pour la réalisation du programme « GenOmics : séquençage à haut débit & maladies rares » visant à élucider les bases génétiques et moléculaires des maladies rares en s’appuyant sur le séquençage à haut débit.
Notre plateforme a bénéficié d’un soutien financier de nombreux partenaires pour l’acquisition d’équipements et de technologies nécessaires à son développement et pour le financement de personnels, que nous remercions.

Nouvelles applications et technologies
Au cours des dernières années, nous avons acquis une solide expérience dans les technologies de séquençage pour cellule unique (prix « Expertises Recherche » 2021 de l’Université de Strasbourg). Nous continuons à implémenter de nouvelles applications chaque année. En parallèle, nous travaillons à la mise en œuvre de deux technologies de transcriptomique spatiale complémentaires, les technologies Visium de 10X Genomics et GeoMx de NanoString.
Publications
2011
Article dans une revue
Helios Is Associated with CD4 T Cells Differentiating to T Helper 2 and Follicular Helper T Cells In Vivo Independently of Foxp3 Expression
- Karine Serre
- Cécile Bénézech
- Guillaume Desanti
- Saeeda Bobat
- Kai-Michael Toellner
- Roger Bird
- Susan Chan
- Philippe Kastner
- Adam F. Cunningham
- Ian C. M. Maclennan
- Elodie Mohr
PLoS ONE ; Volume: 6 ; Page: e20731
2010
Article dans une revue
Myocytic Androgen Receptor Controls the Strength but Not the Mass of Limb Muscles
- Céline Chambon
- Delphine Duteil
- Alban Vignaud
- Arnaud Ferry
- Nadia Messaddeq
- Rocco Malivindi
- Shigeaki Kato
- Pierre Chambon
- Daniel Metzger
Proceedings of the National Academy of Sciences of the United States of America ; Volume: 107 ; Page: 14327--14332
2009
Article dans une revue
164th ENMC International workshop: 6th workshop on centronuclear (myotubular) myopathies, 16-18th January 2009, Naarden, The Netherlands
- Heinz Jungbluth
- Carina Wallgren-Pettersson
- Jocelyn Laporte
Neuromuscul Disord ; Volume: 19 ; Page: 721-729
Article dans une revue
Tailoring carbon nanotube surfaces with glyconanorings: new bionanomaterials with specific lectin affinity.
- Noureddine Khiar
- Manuel Pernia Leal
- Rachid Baati
- Christine Ruhlmann
- Charles Mioskowski
- Patrick Schultz
- Inmaculada Fernández
Chemical Communications ; Page: 4121-3
Article dans une revue
Efficient temporally-controlled targeted mutagenesis in smooth muscle cells of the adult mouse.
- Olivia Wendling
- Jean-Marc Bornert
- Pierre Chambon
- Daniel Metzger
Genesis - The Journal of Genetics and Development ; Volume: 47 ; Page: 14-8
2008
Article dans une revue
The Coffin-Lowry syndrome-associated protein RSK2 is implicated in calcium-regulated exocytosis through the regulation of PLD1.
- Maria Zeniou-Meyer
- Yuanyuan Liu
- Aurélie Béglé
- Mary Olanish
- André Hanauer
- Ute Becherer
- Jens Rettig
- Marie-France Bader
- Nicolas Vitale
Proceedings of the National Academy of Sciences of the United States of America ; Volume: 105 ; Page: 8434-9
Article dans une revue
Temporally controlled ablation of PTEN in adult mouse prostate epithelium generates a model of invasive prostatic adenocarcinoma.
- Chandrahas Koumar Ratnacaram
- Marius Teletin
- Ming Jiang
- Xiangjun Meng
- Pierre Chambon
- Daniel Metzger
Proceedings of the National Academy of Sciences of the United States of America ; Volume: 105 ; Page: 2521-6
2007
Article dans une revue
Critical Role of Desolvation in the Binding of 20-Hydroxyecdysone to the Ecdysone Receptor.
- Christopher Browning
- Elyette Martin
- Caroline Loch
- Jean-Marie Wurtz
- Dino Moras
- Roland H Stote
- Annick P Dejaegere
- Isabelle M L Billas
Journal of Biological Chemistry ; Volume: 282 ; Page: 32924-32934
Article dans une revue
A new form of childhood onset, autosomal recessive spinocerebellar ataxia and epilepsy is localized at 16q21-q23.
- M. Gribaa
- M. Salih
- Mathieu Anheim
- C. Lagier-Tourenne
- D. H'Mida
- N. Drouot
- A. Mohamed
- S. Elmalik
- M. Kabiraj
- M. Al-Rayess
- M. Almubarak
- C. Bétard
- H. Goebel
- M. Koenig
Brain - A Journal of Neurology ; Volume: 130 ; Page: 1921-8
Article dans une revue
Spectrum of NPHP6/CEP290 mutations in Leber congenital amaurosis and delineation of the associated phenotype.
- Isabelle Perrault
- Nathalie Delphin
- Sylvain Hanein
- Sylvie Gerber
- Jean-Louis Dufier
- Olivier Roche
- Sabine Defoort-Dhellemmes
- Hélène Dollfus
- Elisa Fazzi
- Arnold Munnich
- Josseline Kaplan
- Jean-Michel Rozet
Human Mutation ; Volume: 28 ; Page: 416

