
GenomEast
GenomEast
RESPONSABLE DE PLATEFORME
Forts d'une expérience de plus de 20 ans, nous proposons une large gamme de services pour explorer les génomes, leur expression et leur régulation, du contrôle qualité des échantillons de départ jusqu'à l'analyse des données. Ces services sont destinés à l'ensemble de la communauté scientifique, nationale et internationale, publique et privée.



La plateforme est membre du noyau central de l'Infrastructure Nationale de Recherche France Génomique.
La plateforme est labellisée IBISA.
La plateforme est certifiée ISO 9001 - NF X 50-900.
Nos prestations
Nos prestations et nos recommandations sont détaillées dans les fiches produits ci-contre.
Actuellement équipés des séquenceur NextSeq 2000 Illumina et DNBSEQ-G400 de MGI, nous réalisons des projets d'analyse :
du transcriptome :
- RNA-seq
- Single cell RNA-seq (scRNA-seq)
- Transcriptomique spatiale
de l'épigénome :
- ChIP-seq
- Cut & Run
- MeDIP-seq
- Nous avons également de l'expérience dans le séquençage de vos librairies ATAC-seq ou Cut & Tag
du génome :
- Régions ciblées du génome (exome, régions d'intérêts)
- Séquençage complet du génome
multiomique :
- Expression des gènes + ATAC en cellule unique
Pour ces projets, nous préparons et séquençons vos librairies en réalisant des contrôles qualité à chaque étape. Nous pouvons également réaliser l’analyse bioinformatique de vos données. Nous avons une expertise dans une grande variété d’applications et nous avons développé des pipelines bioinformatiques dédiés pour analyser les résultats correspondants.
Alternativement, vous pouvez utiliser notre capacité de séquençage pour séquencer vos propres libraires dans la mesure où elles sont compatibles avec la technologie Illumina.
Fiches produits
- Transcriptomique
- Epigénomique
Multiomique
- Génomique
Besoin d'aide pour le design de votre expérience ?
Consultez nos fiches produit ci-dessus ou nos schémas d’aide à la décision ci-après :
Librairies
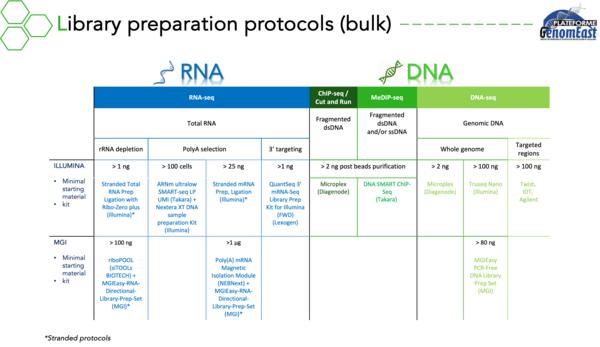
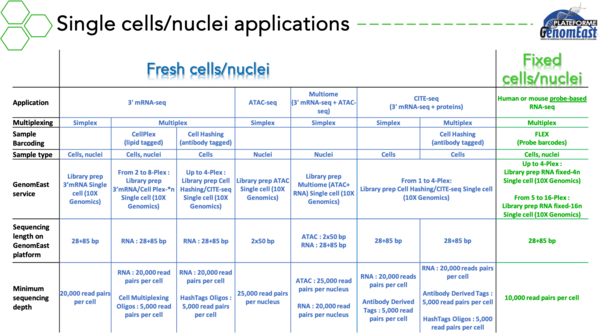
Cellules / Noyaux uniques
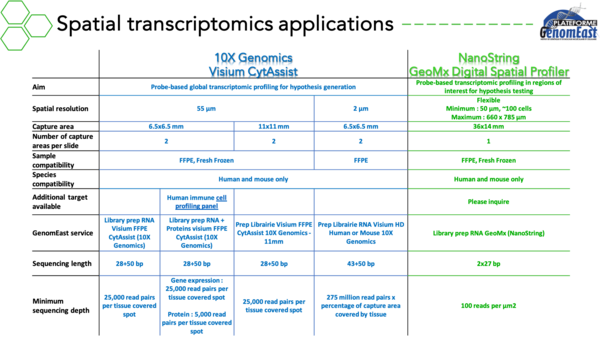
Transcriptomique spatiale
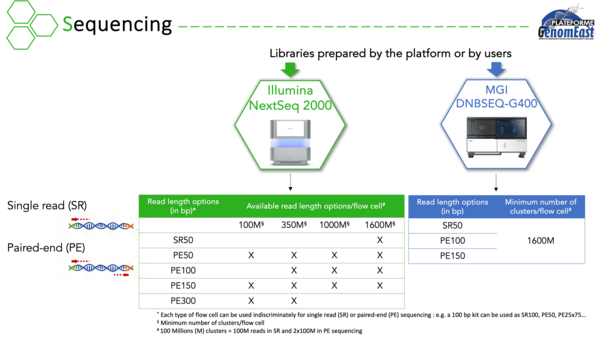
Séquençage
Des questions ?
N’hésitez pas à nous contacter (prenom.nom@igbmc.fr) :
Pour les projets portant sur l’ARN : Christelle Thibault et Céline Keime
Pour les projets portant sur l’ADN : Bernard Jost et Tao Ye
Soumettre un projet
Comment soumettre un projet à GenomEast ?
Via notre interface web
Quelle procédure suivre ?
Comment envoyer des échantillons à GenomEast ?
Tous nos services sont régis par les conditions générales de vente qui prévalent sur toutes les conditions d'achat, sauf accord écrit de notre part. Vous pouvez également consulter notre plan de gestion des données.
Formations
Nous partageons régulièrement nos connaissances et expertises à travers différents enseignements et formations, notamment :
L'UE « High Throughput Approaches » à l’Ecole Supérieure de Biotechnologie de Strasbourg (ESBS)
L’UE « Omique 2 » du Master 2 Biologie-Santé, parcours Recherche en Biomédecine à l’Université de Strasbourg
Le PhD Program de l’école Universitaire de Recherche IMCBio à Strasbourg
Le DU « Séquençage haut débit et maladies rares » à l'Université de Dijon
L'école de bioinformatique AVIESAN IFB Inserm
L’organisation de formations en bioinformatique pour le séquençage haut débit avec l’Inserm et le CNRS
Membres
Responsable de la plateforme
Christelle Thibault-Carpentier
Traitement des échantillons
- Bernard Jost (responsable)
- Marie Cerciat
- Amal El Amri
- Romain Kaiser
- Christelle Morgenthaler-Roth
- Angélique Pichot
- David Rodriguez
- Serge Vicaire
Bioinformatique
- Céline Keime (responsable)
- Matthieu Jung
- Stéphanie Le Gras
- Damien Plassard
- Tao Ye
Ressources
La plateforme est équipée de technologies de pointes parmis lesquelles :

Le séquenceur haut débit NextSeq 2000 (Illumina)

Le séquenceur haut débit AVITI (Element Biosciences)

La technologie en cellule unique du Chromium iX (10x Genomics)

La technologie spatial Visium CytAssist (10X Genomics)
Nos autres équipements:
- Séquençage haut débit : iSeq100 (Illumina)
- Transcriptomique spatiale : GeoMx Digital Spatial Profiler (NanoString)
- Contrôle qualité : Flash Reader Varioskan et Qubit Fluorometer (Thermo Fisher), 2100 Bioanalyzer et Fragment Analyzer AATI (Agilent)
- Sonicateur : E220 AFA system (Covaris)
- Infrastructure informatique : calcul (Dell) : 592 cœurs, stockage (Lenovo, GPFS) : 750 To
Réseaux et financements
France Génomique
Notre plateforme est partenaire du réseau France Génomique depuis la sélection initiale du projet dans le cadre des « Investissements d'avenir » en 2011. Ce réseau rassemble et partage les ressources des principales plateformes françaises de génomique et de bioinformatique.
Fondation maladies rares
Depuis 2011, nous sommes l’une des plateformes partenaires de la Fondation Maladies Rares pour la réalisation du programme « GenOmics : séquençage à haut débit & maladies rares » visant à élucider les bases génétiques et moléculaires des maladies rares en s’appuyant sur le séquençage à haut débit.
Notre plateforme a bénéficié d’un soutien financier de nombreux partenaires pour l’acquisition d’équipements et de technologies nécessaires à son développement et pour le financement de personnels, que nous remercions.

Nouvelles applications et technologies
Au cours des dernières années, nous avons acquis une solide expérience dans les technologies de séquençage pour cellule unique (prix « Expertises Recherche » 2021 de l’Université de Strasbourg). Nous continuons à implémenter de nouvelles applications chaque année. En parallèle, nous travaillons à la mise en œuvre de deux technologies de transcriptomique spatiale complémentaires, les technologies Visium de 10X Genomics et GeoMx de NanoString.
Publications
2006
Article dans une revue
Prepubertal testis development relies on retinoic acid but not rexinoid receptors in Sertoli cells.
- Nadège Vernet
- Christine Dennefeld
- Florian Guillou
- Pierre Chambon
- Norbert B Ghyselinck
- Manuel Mark
EMBO Journal ; Volume: 25 ; Page: 5816-25
Article dans une revue
Primary tumour genetic alterations and intra-tumoral heterogeneity are maintained in xenografts of human colon cancers showing chromosome instability.
- D. Guenot
- E. Guérin
- S. Aguillon-Romain
- E. Pencreach
- A. Schneider
- A. Neuville
- M.-P. Chenard
- I. Duluc
- S. Du Manoir
- C. Brigand
- P. Oudet
- M. Kedinger
- M.-P. Gaub
Journal of Pathology ; Volume: 208 ; Page: 643-52
Chapitre d’ouvrage
Mouse Models for Friedreich's Ataxia
- Hélène Puccio
Genetic Instabilities and Neurological Diseases (Second Edition) ; Page: 321-326
2005
Article dans une revue
The Natural Mutation Encoding a C Terminus-Truncated 5-Hydroxytryptamine2B Receptor Is a Gain of Proliferative Functions
- Maud Deraet
- Philippe Manivet
- Agnes Janoshazi
- Jacques Callebert
- Silke Guenther
- Ludovic Drouet
- Jean-Marie Launay
- Luc Maroteaux
Molecular Pharmacology ; Volume: 67 ; Page: 983 - 991
Article dans une revue
Cloning, purification and crystallization of a Walker-type Pyrococcus abyssi ATPase family member
- Muriel Uhring
- Gilbert Bey
- Odile Lecompte
- Jean Cavarelli
- Dino Moras
- Olivier Poch
Acta Crystallographica Section F: Structural Biology and Crystallization Communications ; Volume: 61 ; Page: 925-927
2004
Article dans une revue
Monitoring ligand-mediated nuclear receptor-coregulator interactions by noncovalent mass spectrometry
- Sarah Sanglier
- William Bourguet
- Pierre Germain
- Virginie Chavant
- Dino Moras
- Hinrich Gronemeyer
- Noelle Potier
- Alain van Dorsselaer
European Journal of Biochemistry ; Volume: 271 ; Page: 4958-4967
Article dans une revue
Expression of the RSK2 gene during early human development
- Fabien Guimiot
- Anne-Lise Delezoide
- Andre Hanauer
- Michel Simonneau
Gene Expression Patterns ; Volume: 4 ; Page: 111-114
2003
Article dans une revue
Myotubularins, a large disease-associated family of cooperating catalytically active and inactive phosphoinositides phosphatases
- Jocelyn Laporte
- Florence Bedez
- Alessandra Bolino
- Jean-Louis Mandel
Human Molecular Genetics ; Volume: 12 ; Page: R285-R292
Article dans une revue
Tamoxifen-inducible glia-specific Cre mice for somatic mutagenesis in oligodendrocytes and Schwann cells
- Dino P. Leone
- Stéphane Genoud
- Suzana Atanasoski
- Reinhard Grausenburger
- Philipp Berger
- Daniel Metzger
- Wendy B. Macklin
- Pierre Chambon
- Ueli Suter
Molecular and Cellular Neuroscience ; Volume: 22 ; Page: 430-440
Article dans une revue
Insulin and estrogen receptor ligand influence the FGF-2 activities in MCF-7 breast cancer cells.
- Martine Garnier
- Claire Giamarchi
- Isabelle Delrieu
- Marie-Christine Rio
- Patrick Chinestra
- Francis Bayard
- Marc E. Poirot
- Jean-Charles Faye
Biochemical Pharmacology ; Volume: 65 ; Page: 629-636

