
GenomEast
GenomEast
RESPONSABLE DE PLATEFORME
Forts d'une expérience de plus de 20 ans, nous proposons une large gamme de services pour explorer les génomes, leur expression et leur régulation, du contrôle qualité des échantillons de départ jusqu'à l'analyse des données. Ces services sont destinés à l'ensemble de la communauté scientifique, nationale et internationale, publique et privée.



La plateforme est membre du noyau central de l'Infrastructure Nationale de Recherche France Génomique.
La plateforme est labellisée IBISA.
La plateforme est certifiée ISO 9001 - NF X 50-900.
Nos prestations
Nos prestations et nos recommandations sont détaillées dans les fiches produits ci-contre.
Actuellement équipés des séquenceur NextSeq 2000 Illumina et DNBSEQ-G400 de MGI, nous réalisons des projets d'analyse :
du transcriptome :
- RNA-seq
- Single cell RNA-seq (scRNA-seq)
- Transcriptomique spatiale
de l'épigénome :
- ChIP-seq
- Cut & Run
- MeDIP-seq
- Nous avons également de l'expérience dans le séquençage de vos librairies ATAC-seq ou Cut & Tag
du génome :
- Régions ciblées du génome (exome, régions d'intérêts)
- Séquençage complet du génome
multiomique :
- Expression des gènes + ATAC en cellule unique
Pour ces projets, nous préparons et séquençons vos librairies en réalisant des contrôles qualité à chaque étape. Nous pouvons également réaliser l’analyse bioinformatique de vos données. Nous avons une expertise dans une grande variété d’applications et nous avons développé des pipelines bioinformatiques dédiés pour analyser les résultats correspondants.
Alternativement, vous pouvez utiliser notre capacité de séquençage pour séquencer vos propres libraires dans la mesure où elles sont compatibles avec la technologie Illumina.
Fiches produits
- Transcriptomique
- Epigénomique
Multiomique
- Génomique
Besoin d'aide pour le design de votre expérience ?
Consultez nos fiches produit ci-dessus ou nos schémas d’aide à la décision ci-après :
Librairies
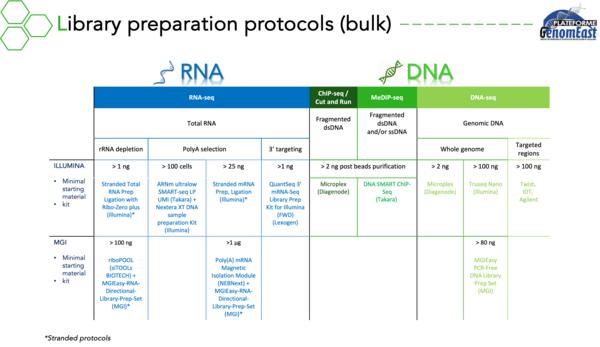
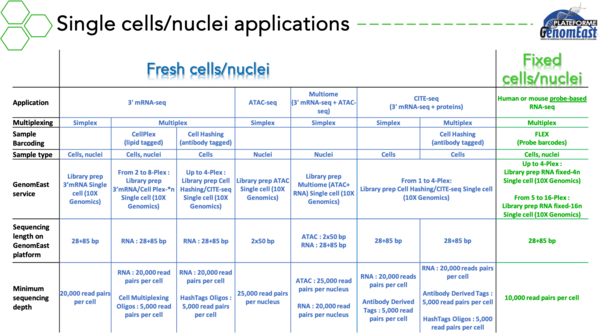
Cellules / Noyaux uniques
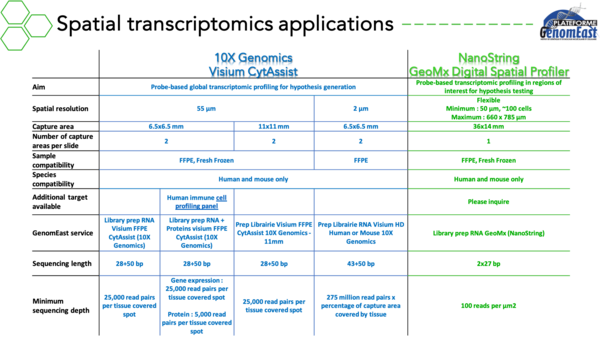
Transcriptomique spatiale
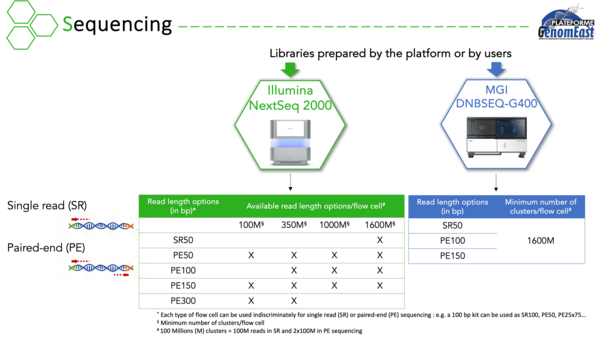
Séquençage
Des questions ?
N’hésitez pas à nous contacter (prenom.nom@igbmc.fr) :
Pour les projets portant sur l’ARN : Christelle Thibault et Céline Keime
Pour les projets portant sur l’ADN : Bernard Jost et Tao Ye
Soumettre un projet
Comment soumettre un projet à GenomEast ?
Via notre interface web
Quelle procédure suivre ?
Comment envoyer des échantillons à GenomEast ?
Tous nos services sont régis par les conditions générales de vente qui prévalent sur toutes les conditions d'achat, sauf accord écrit de notre part. Vous pouvez également consulter notre plan de gestion des données.
Formations
Nous partageons régulièrement nos connaissances et expertises à travers différents enseignements et formations, notamment :
L'UE « High Throughput Approaches » à l’Ecole Supérieure de Biotechnologie de Strasbourg (ESBS)
L’UE « Omique 2 » du Master 2 Biologie-Santé, parcours Recherche en Biomédecine à l’Université de Strasbourg
Le PhD Program de l’école Universitaire de Recherche IMCBio à Strasbourg
Le DU « Séquençage haut débit et maladies rares » à l'Université de Dijon
L'école de bioinformatique AVIESAN IFB Inserm
L’organisation de formations en bioinformatique pour le séquençage haut débit avec l’Inserm et le CNRS
Membres
Responsable de la plateforme
Christelle Thibault-Carpentier
Traitement des échantillons
- Bernard Jost (responsable)
- Marie Cerciat
- Amal El Amri
- Romain Kaiser
- Christelle Morgenthaler-Roth
- Angélique Pichot
- David Rodriguez
- Serge Vicaire
Bioinformatique
- Céline Keime (responsable)
- Matthieu Jung
- Stéphanie Le Gras
- Damien Plassard
- Tao Ye
Ressources
La plateforme est équipée de technologies de pointes parmis lesquelles :

Le séquenceur haut débit NextSeq 2000 (Illumina)

Le séquenceur haut débit AVITI (Element Biosciences)

La technologie en cellule unique du Chromium iX (10x Genomics)

La technologie spatial Visium CytAssist (10X Genomics)
Nos autres équipements:
- Séquençage haut débit : iSeq100 (Illumina)
- Transcriptomique spatiale : GeoMx Digital Spatial Profiler (NanoString)
- Contrôle qualité : Flash Reader Varioskan et Qubit Fluorometer (Thermo Fisher), 2100 Bioanalyzer et Fragment Analyzer AATI (Agilent)
- Sonicateur : E220 AFA system (Covaris)
- Infrastructure informatique : calcul (Dell) : 592 cœurs, stockage (Lenovo, GPFS) : 750 To
Réseaux et financements
France Génomique
Notre plateforme est partenaire du réseau France Génomique depuis la sélection initiale du projet dans le cadre des « Investissements d'avenir » en 2011. Ce réseau rassemble et partage les ressources des principales plateformes françaises de génomique et de bioinformatique.
Fondation maladies rares
Depuis 2011, nous sommes l’une des plateformes partenaires de la Fondation Maladies Rares pour la réalisation du programme « GenOmics : séquençage à haut débit & maladies rares » visant à élucider les bases génétiques et moléculaires des maladies rares en s’appuyant sur le séquençage à haut débit.
Notre plateforme a bénéficié d’un soutien financier de nombreux partenaires pour l’acquisition d’équipements et de technologies nécessaires à son développement et pour le financement de personnels, que nous remercions.

Nouvelles applications et technologies
Au cours des dernières années, nous avons acquis une solide expérience dans les technologies de séquençage pour cellule unique (prix « Expertises Recherche » 2021 de l’Université de Strasbourg). Nous continuons à implémenter de nouvelles applications chaque année. En parallèle, nous travaillons à la mise en œuvre de deux technologies de transcriptomique spatiale complémentaires, les technologies Visium de 10X Genomics et GeoMx de NanoString.
Publications
2019
Article dans une revue
A genome‐wide screen identifies IRF2 as a key regulator of caspase‐4 in human cells
- Sacha Benaoudia
- Amandine Martin
- Marta Puig Gamez
- Gabrielle Gay
- Brice Lagrange
- Maxence Cornut
- Kyrylo Krasnykov
- Jean‐baptiste Claude
- Cyril Bourgeois
- Sandrine Hughes
- Benjamin Gillet
- Omran Allatif
- Antoine Corbin
- Romeo Ricci
- Thomas Henry
EMBO Reports ; Volume: 20
Article dans une revue
Transcriptional Regulation of the Glutamate/GABA/Glutamine Cycle in Adult Glia Controls Motor Activity and Seizures in Drosophila
- David Mazaud
- Benjamin Kottler
- Catarina Gonçalves-Pimentel
- Sandra Proelss
- Nadine Tüchler
- Celine Deneubourg
- Yoshihiro Yuasa
- Celine Diebold
- Heinz Jungbluth
- Eric C Lai
- Frank Hirth
- Angela Giangrande
- Manolis Fanto
Journal of Neuroscience ; Volume: 39 ; Page: 5269-5283
Article dans une revue
Mu opioid receptors in the medial habenula contribute to naloxone aversion
- Laura-Joy Boulos
- Sami Ben Hamida
- Julie Bailly
- Malosree Maitra
- Aliza Toby Ehrlich
- Claire Gaveriaux-Ruff
- Emmanuel Darcq
- Brigitte Kieffer
Neuropsychopharmacology ; Volume: 45 ; Page: 247-255
Article dans une revue
GLRX5 mutations impair heme biosynthetic enzymes ALA synthase 2 and ferrochelatase in Human congenital sideroblastic anemia
- Raed Daher
- Abdellah Mansouri
- Alain Martelli
- Sophie Bayart
- Hana Manceau
- Isabelle Callebaut
- Boualem Moulouel
- Laurent Gouya
- Hervé Puy
- Caroline Kannengiesser
- Zoubida Karim
Molecular Genetics and Metabolism
2017
Article dans une revue
Synthesis facilitates an understanding of the structural basis for translation inhibition by the lissoclimides
- Zef Könst
- Anne Szklarski
- Simone Pellegrino
- Sharon Michalak
- Mélanie Meyer
- Camila Zanette
- Regina Cencic
- Sangkil Nam
- Vamsee Voora
- David Horne
- Jerry Pelletier
- David Mobley
- Gulnara Yusupova
- Marat Yusupov
- Christopher Vanderwal
Nature Chemistry ; Volume: 9 ; Page: 1140-1149
Article dans une revue
The Ribosome Holds the RNA Polymerase on Track in Bacteria
- Bruno P. Klaholz
Trends in Biochemical Sciences ; Volume: 42 ; Page: 686-689
Article dans une revue
Rare GABRA3 variants are associated with epileptic seizures, encephalopathy and dysmorphic features
- Cristina Niturad
- Dorit Lev
- Vera Kalscheuer
- Agnieszka Charzewska
- Julian Schubert
- Tally Lerman-Sagie
- Hester Kroes
- Renske Oegema
- Monica Traverso
- Nicola Specchio
- Maria Lassota
- Jameleddine Chelly
- Odeya Bennett-Back
- Nirit Carmi
- Tal Koffler-Brill
- Michele Iacomino
- Marina Trivisano
- Giuseppe Capovilla
- Pasquale Striano
- Magdalena Nawara
- ...
Brain - A Journal of Neurology ; Volume: 140 ; Page: 2879-2894
2016
Article dans une revue
Solution structure of the 5'-terminal hairpin of the 7SK small nuclear RNA
- Sarah Bourbigot
- Anne-Catherine Dock-Bregeon
- Pascal Eberling
- Jérôme Coutant
- Bruno Kieffer
- Isabelle Lebars
RNA ; Volume: 22 ; Page: 1844-1858
Article dans une revue
Epidermal-specific deletion of CD44 reveals a function in keratinocytes in response to mechanical stress
- M. Shatirishvili
- A. S. Burk
- C. M. Franz
- G. Pace
- T. Kastilan
- K. Breuhahn
- E. Hinterseer
- Andree Dierich
- L. Bakiri
- E. F. Wagner
- H. Ponta
- T. N. Hartmann
- M. Tanaka
- V. Orian-Rousseau
Cell Death and Disease ; Volume: 7
Article dans une revue
Myelinosomes act as natural secretory organelles in Sertoli cells to prevent accumulation of aggregate-prone mutant Huntingtin and CFTR
- Marina Yefimova
- Emile Béré
- Anne Cantereau-Becq
- Thomas Harnois
- Annie-Claire Meunier
- Nadia Messaddeq
- Frédéric Becq
- Yvon Trottier
- Nicolas Bourmeyster
Human Molecular Genetics ; Volume: 25 ; Page: 4170-4185

