
GenomEast
GenomEast
RESPONSABLE DE PLATEFORME
Forts d'une expérience de plus de 20 ans, nous proposons une large gamme de services pour explorer les génomes, leur expression et leur régulation, du contrôle qualité des échantillons de départ jusqu'à l'analyse des données. Ces services sont destinés à l'ensemble de la communauté scientifique, nationale et internationale, publique et privée.



La plateforme est membre du noyau central de l'Infrastructure Nationale de Recherche France Génomique.
La plateforme est labellisée IBISA.
La plateforme est certifiée ISO 9001 - NF X 50-900.
Nos prestations
Nos prestations et nos recommandations sont détaillées dans les fiches produits ci-contre.
Actuellement équipés des séquenceur NextSeq 2000 Illumina et DNBSEQ-G400 de MGI, nous réalisons des projets d'analyse :
du transcriptome :
- RNA-seq
- Single cell RNA-seq (scRNA-seq)
- Transcriptomique spatiale
de l'épigénome :
- ChIP-seq
- Cut & Run
- MeDIP-seq
- Nous avons également de l'expérience dans le séquençage de vos librairies ATAC-seq ou Cut & Tag
du génome :
- Régions ciblées du génome (exome, régions d'intérêts)
- Séquençage complet du génome
multiomique :
- Expression des gènes + ATAC en cellule unique
Pour ces projets, nous préparons et séquençons vos librairies en réalisant des contrôles qualité à chaque étape. Nous pouvons également réaliser l’analyse bioinformatique de vos données. Nous avons une expertise dans une grande variété d’applications et nous avons développé des pipelines bioinformatiques dédiés pour analyser les résultats correspondants.
Alternativement, vous pouvez utiliser notre capacité de séquençage pour séquencer vos propres libraires dans la mesure où elles sont compatibles avec la technologie Illumina.
Fiches produits
- Transcriptomique
- Epigénomique
Multiomique
- Génomique
Besoin d'aide pour le design de votre expérience ?
Consultez nos fiches produit ci-dessus ou nos schémas d’aide à la décision ci-après :
Librairies
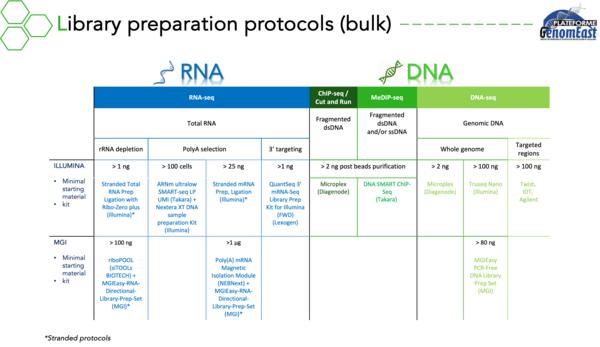
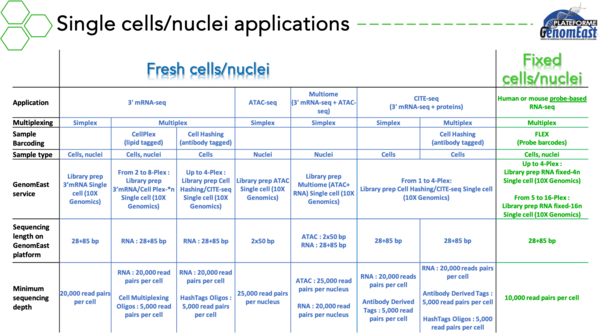
Cellules / Noyaux uniques
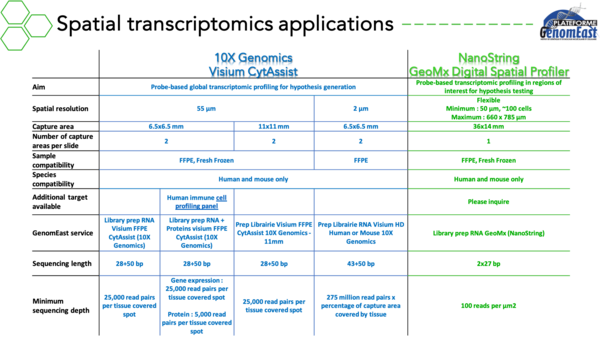
Transcriptomique spatiale
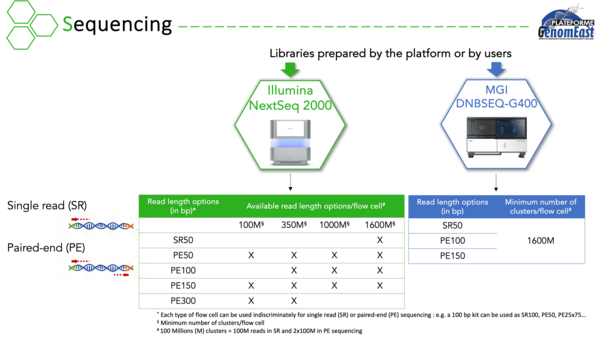
Séquençage
Des questions ?
N’hésitez pas à nous contacter (prenom.nom@igbmc.fr) :
Pour les projets portant sur l’ARN : Christelle Thibault et Céline Keime
Pour les projets portant sur l’ADN : Bernard Jost et Tao Ye
Soumettre un projet
Comment soumettre un projet à GenomEast ?
Via notre interface web
Quelle procédure suivre ?
Comment envoyer des échantillons à GenomEast ?
Tous nos services sont régis par les conditions générales de vente qui prévalent sur toutes les conditions d'achat, sauf accord écrit de notre part. Vous pouvez également consulter notre plan de gestion des données.
Formations
Nous partageons régulièrement nos connaissances et expertises à travers différents enseignements et formations, notamment :
L'UE « High Throughput Approaches » à l’Ecole Supérieure de Biotechnologie de Strasbourg (ESBS)
L’UE « Omique 2 » du Master 2 Biologie-Santé, parcours Recherche en Biomédecine à l’Université de Strasbourg
Le PhD Program de l’école Universitaire de Recherche IMCBio à Strasbourg
Le DU « Séquençage haut débit et maladies rares » à l'Université de Dijon
L'école de bioinformatique AVIESAN IFB Inserm
L’organisation de formations en bioinformatique pour le séquençage haut débit avec l’Inserm et le CNRS
Membres
Responsable de la plateforme
Christelle Thibault-Carpentier
Traitement des échantillons
- Bernard Jost (responsable)
- Marie Cerciat
- Amal El Amri
- Romain Kaiser
- Christelle Morgenthaler-Roth
- Angélique Pichot
- David Rodriguez
- Serge Vicaire
Bioinformatique
- Céline Keime (responsable)
- Matthieu Jung
- Stéphanie Le Gras
- Damien Plassard
- Tao Ye
Ressources
La plateforme est équipée de technologies de pointes parmis lesquelles :

Le séquenceur haut débit NextSeq 2000 (Illumina)

Le séquenceur haut débit AVITI (Element Biosciences)

La technologie en cellule unique du Chromium iX (10x Genomics)

La technologie spatial Visium CytAssist (10X Genomics)
Nos autres équipements:
- Séquençage haut débit : iSeq100 (Illumina)
- Transcriptomique spatiale : GeoMx Digital Spatial Profiler (NanoString)
- Contrôle qualité : Flash Reader Varioskan et Qubit Fluorometer (Thermo Fisher), 2100 Bioanalyzer et Fragment Analyzer AATI (Agilent)
- Sonicateur : E220 AFA system (Covaris)
- Infrastructure informatique : calcul (Dell) : 592 cœurs, stockage (Lenovo, GPFS) : 750 To
Réseaux et financements
France Génomique
Notre plateforme est partenaire du réseau France Génomique depuis la sélection initiale du projet dans le cadre des « Investissements d'avenir » en 2011. Ce réseau rassemble et partage les ressources des principales plateformes françaises de génomique et de bioinformatique.
Fondation maladies rares
Depuis 2011, nous sommes l’une des plateformes partenaires de la Fondation Maladies Rares pour la réalisation du programme « GenOmics : séquençage à haut débit & maladies rares » visant à élucider les bases génétiques et moléculaires des maladies rares en s’appuyant sur le séquençage à haut débit.
Notre plateforme a bénéficié d’un soutien financier de nombreux partenaires pour l’acquisition d’équipements et de technologies nécessaires à son développement et pour le financement de personnels, que nous remercions.

Nouvelles applications et technologies
Au cours des dernières années, nous avons acquis une solide expérience dans les technologies de séquençage pour cellule unique (prix « Expertises Recherche » 2021 de l’Université de Strasbourg). Nous continuons à implémenter de nouvelles applications chaque année. En parallèle, nous travaillons à la mise en œuvre de deux technologies de transcriptomique spatiale complémentaires, les technologies Visium de 10X Genomics et GeoMx de NanoString.
Publications
2013
Article dans une revue
Loss of Catalytically Inactive Lipid Phosphatase Myotubularin-related Protein 12 Impairs Myotubularin Stability and Promotes Centronuclear Myopathy in Zebrafish
- Vandana A. Gupta
- Karim Hnia
- Laura L. Smith
- Stacey R. Gundry
- Jessica E. Mcintire
- Junko Shimazu
- Jessica R. Bass
- Ethan A. Talbot
- Leonela Amoasii
- Nathaniel E. Goldman
- Jocelyn Laporte
- Alan H. Beggs
PLoS Genetics ; Volume: 9 ; Page: e1003583
Article dans une revue
Topology of mRNA chain in isolated eukaryotic double-row polyribosomes
- Zh. A. Afonina
- Alexander Myasnikov
- N. F. Khabibullina
- A. Y. Belorusova
- Jean-François Ménétret
- V. D. Vasiliev
- Bruno Klaholz
- V. A. Shirokov
- A. S. Spirin
Biochemistry (mosc.) ; Volume: 78 ; Page: 445-454
2012
Article dans une revue
The nuclear hormone receptor PPARgamma counteracts vascular calcification by inhibiting Wnt5a signalling in vascular smooth muscle cells
- Estelle Woldt
- Jerôme Terrand
- Mohamed Mlih
- Rachel Matz
- Veronique Bruban
- Fanny Coudane
- Sophie Foppolo
- Zeina El Asmar
- M Chollet
- E Ninio
- Audrey Bednarczyk
- Danièle Thiersé
- Christine Schaeffer
- Alain van Dorsselaer
- Christian Boudier
- W Wahli
- Pierre Chambon
- Daniel Metzger
- J Herz
- Philippe Boucher
Nature Communications ; Volume: 3 ; Page: 1077
Article dans une revue
The App-Runx1 Region Is Critical for Birth Defects and Electrocardiographic Dysfunctions Observed in a Down Syndrome Mouse Model
- Matthieu Raveau
- Jacques Lignon
- Valérie Nalesso
- Arnaud Duchon
- Yoram Groner
- Andrew Sharp
- Doulaye Dembele
- Véronique Brault
- Yann Hérault
PLoS Genetics ; Volume: 8 ; Page: e1002724
Article dans une revue
A role for the RNA pol II–associated PAF complex in AID-induced immune diversification
- Katharina L. Willmann
- Sara Milosevic
- Siim Pauklin
- Kerstin-Maike Schmitz
- Gopinath Rangam
- Maria T. Simon
- Sarah Maslen
- Mark Skehel
- Isabelle Robert
- Vincent Heyer
- Ebe Schiavo
- Bernardo Reina-San-Martin
- Svend K. Petersen-Mahrt
Journal of Experimental Medicine ; Volume: 209 ; Page: 2099--2111
Article dans une revue
Unlike for Human Monocytes after LPS Activation, Release of TNF-α by THP-1 Cells Is Produced by a TACE Catalytically Different from Constitutive TACE
- Helena Moreira-Tabaka
- Jean Peluso
- Jean‐luc Vonesch
- Didier Hentsch
- Pascal Kessler
- Jean-Marie Reimund
- Serge Dumont
- Christian D. Muller
PLoS ONE ; Volume: 7 ; Page: e34184
Article dans une revue
Next generation sequencing for molecular diagnosis of neuromuscular diseases
- Nasim Vasli
- Johann Böhm
- Stéphanie Le Gras
- Jean Muller
- Cécile Pizot
- Bernard Jost
- Andoni Echaniz‐laguna
- Vincent Laugel
- Christine Tranchant
- Rafaelle Bernard
- Frederic Plewniak
- Serge Vicaire
- Nicolas Levy
- Jamel Chelly
- Jean-Louis Mandel
- Valerie Biancalana
- Jocelyn Laporte
Acta Neuropathologica ; Volume: 124 ; Page: 273 - 283
Article dans une revue
A unified phylogeny-based nomenclature for histone variants
- Paul Talbert
- Kami Ahmad
- Geneviève Almouzni
- Juan Ausió
- Frederic Berger
- Prem Bhalla
- William Bonner
- Zacheus W. Cande
- Brian Chadwick
- Simon W. Chan
- George A. Cross
- Liwang Cui
- Stefan Dimitrov
- Detlef Doenecke
- José Eirin-López
- Martin Gorovsky
- Sandra Hake
- Barbara Hamkalo
- Sarah Holec
- Steven Jacobsen
- ...
Epigenetics & Chromatin ; Volume: 5 ; Page: 7
2011
Article dans une revue
Strategies for bacterial expression of protein–peptide complexes: Application to solubilization of papillomavirus E6
- Abdellahi Ould M’hamed Ould Sidi
- Khaled Ould Babah
- Nicole Brimer
- Yves Nominé
- Christophe Romier
- Bruno Kieffer
- Scott Vande Pol
- Gilles Travé
- Katia Zanier
Protein Expression and Purification ; Volume: 80 ; Page: 8-16
Article dans une revue
PAT-12, a potential anti-nematode target, is a new spectraplakin partner essential for Caenorhabditis elegans hemidesmosome integrity and embryonic morphogenesis
- Suzannah Hetherington
- Christelle Gally
- Julie-Anne Fritz
- Jolanta Polanowska
- Jérôme Reboul
- Yannick Schwab
- Hala Zahreddine
- Carolyn Behm
- Michel Labouesse
Developmental Biology ; Volume: 350 ; Page: 267--278

